Les nouvelles techniques / méthodes du systèmes CRISPR/CAS9

Léo CARRILLO
La découverte du système CRISPR-Cas9 a été une véritable révolution pour la biologie moléculaire et le génie génétique. Ce système a permis des avancées dans de très nombreux domaines. Nous présentons ici les dernières avancées du système en lui-même et comment il a été amélioré au cours des dernières années pour pallier ses principaux défauts.
Introduction
En 2012 Emmanuelle Charpentier, Jennifer Doudna et leurs collègues étudient les mécanismes de défense des bactéries face aux infections et font une découverte qui est aujourd’hui vue comme une révolution pour la biologie moléculaire : le système CRISPR-Cas9. Un article sur le site présente l’histoire de cette découverte et en quoi il est totalement en rupture avec les outils utilisés précédemment.
Depuis sa caractérisation, de très nombreux laboratoires se sont intéressés à cet outil puissant. Pour beaucoup cela représentait un système versatile qui pouvait remplacer les méthodes habituelles d’ingénierie génétique, souvent plus chères et moins efficaces. La technologie CRISPR-Cas9 a commencé à être de plus en plus utilisée pour couper de manière précise et remplacer des séquences d’ADN avec une grande rapidité et efficacité. Les coûts d’utilisation ont rapidement diminué et de nombreux laboratoires se sont intéressés à cet outil pour bien des applications et des domaines différents.
Certains laboratoires se sont quant à eux penchés sur l’outil en lui-même et ont essayé de l’améliorer ou de s’en inspirer pour créer de nouveaux outils de biologie moléculaire innovants et répondant à des problématiques. L’un des inconvénients les plus importants du système CRISPR-Cas9 est qu’il passe par une étape de cassure double brin de l’ADN au niveau de la séquence cible. Une fois la cassure effectuée, la cellule peut utiliser d’autres modèles que l’ARN guide du système pour la réparer, comme par exemple le chromosome homologue. Ceci réduit l’efficacité potentielle de CRISPR-Cas9. La cassure double brin peut également entraîner de plus larges recombinaisons et réarrangements chromosomiques non désirés (Brunner et al., 2019). La plupart des nouvelles méthodes recherchées tentent donc de pallier ce problème majeur pour réduire les désavantages du système.
1- DeadCas9
Une des premières variantes de CRISPR-Cas9 avait pour but de réguler l’expression d’un gène plutôt que de le modifier. Un mutant de la protéine Cas9 a été découvert en 2015, la deadCas9 (dCas9) (Jinek et al., 2012). Ce mutant a la particularité de ne pas avoir d’activité endonucléase qui lui confère habituellement sa capacité à couper un des brins de l’ADN cible. Couplée à un ARN guide dirigé contre une séquence d’un gène ciblé, la protéine Cas9 se fixe au brin complémentaire de l’ARN guide de la même manière que la version non mutée. Cette liaison seule suffit à interférer avec la machinerie de transcription et ainsi diminuer l’expression d’un gène à l’échelle du génome entier (Qi et al., 2013). Cette méthode permet donc de réguler l’expression d’un gène sans changer la séquence de celui-ci et est donc une régulation épigénétique.
La cassure double brin de l’ADN par le système CRISPR-Cas9 classique étant connue pour provoquer beaucoup de modifications non désirées par la machinerie de réparation de la cellule, cette méthode permet donc d’obtenir une régulation d’un gène ciblé avec moins d’effets secondaires.
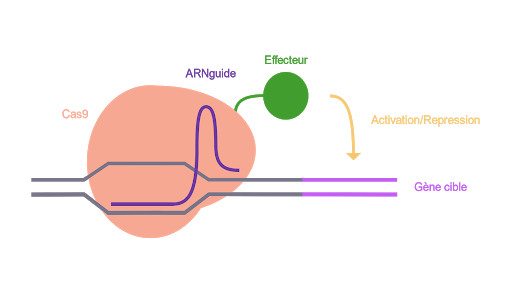
La dCas9 peut également être utilisée en parallèle d’activateurs ou de répresseurs de l’expression des gènes pour les réguler. Une dCas9 peut par exemple être fusionnée avec des domaines ou protéines activateurs transcriptionnels. Le domaine Cas9 de cette protéine de fusion va reconnaître l’ADN cible grâce à l’ARNguide, le domaine d’activation sera ainsi physiquement proche du gène cible et pourra ainsi augmenter son expression selon son mode d’action. Cette approche peut également être employée avec des répresseurs de l’expression des gènes pour augmenter l’efficacité de la seule présence de la Cas9 devant le gène cible. (Fig 1.)
2- Single base editing
Comme mentionné précédemment, la cassure double brin induit de nombreux effets non voulus et peuvent interférer avec les modifications souhaitées sur le gène cible. La majorité des pathologies humaines est causée par des mutations ponctuelles (58% selon la base de donnés ClinVar (Landrum et al., 2014)). Ceci a poussé les chercheurs à trouver un moyen plus efficace de modifier une seule base d’un gène cible, sans avoir recours à des cassures doubles brins, ni à la recombinaison de plusieurs dizaines de nucléotides. Des méthodes dites de “Base Editing” ont ainsi été mise au point. Deux méthodes existent à ce jour. La première, “Cytosine Base Editor”, permet de modifier une cytosine en thymine en désaminant la cytosine cible et en inhibant les voies qui permettraient de réparer un mésappariement. La seconde, “Adenine Base Editor”, permet de modifier une adénine en guanine, en désaminant l’adénine cible. En modifiant l’un ou l’autre brin du gène cible on peut donc créer 4 mutations : C vers T donnant G vers A sur l’autre brin, et A vers G donnant T vers C sur l’autre brin. Ces 4 mutations ponctuelles différentes (sur les 12 possibles) représentent 61% des mutations ponctuelles impliquées dans des pathologies humaines (Landrum et al., 2014).
3 - Prime editing
Enfin une méthode a été développée très récemment pour réécrire entièrement une séquence cible de plusieurs nucléotides sans avoir recours à une cassure double brin ni à de la recombinaison homologue (Anzalone et al., 2019). Cette technique, appelée Prime Editing permet de chercher une séquence spécifique et de la remplacer par une autre séquence voulue. Cette méthode se base sur un ARN contenant plusieurs parties ainsi qu’une protéine de fusion. L’ARN est constitué d’une partie se liant à l’ADN cible et d’une contenant la séquence qui servira de modèle pour réécrire la séquence souhaitée. La protéine quant à elle contient un domaine de la Cas9 qui permet d’ouvrir l’ADN ainsi qu’un domaine de rétrotranscription qui permet de remplacer la séquence cible en recopiant la séquence de l’ARN guide (Fig 2.).
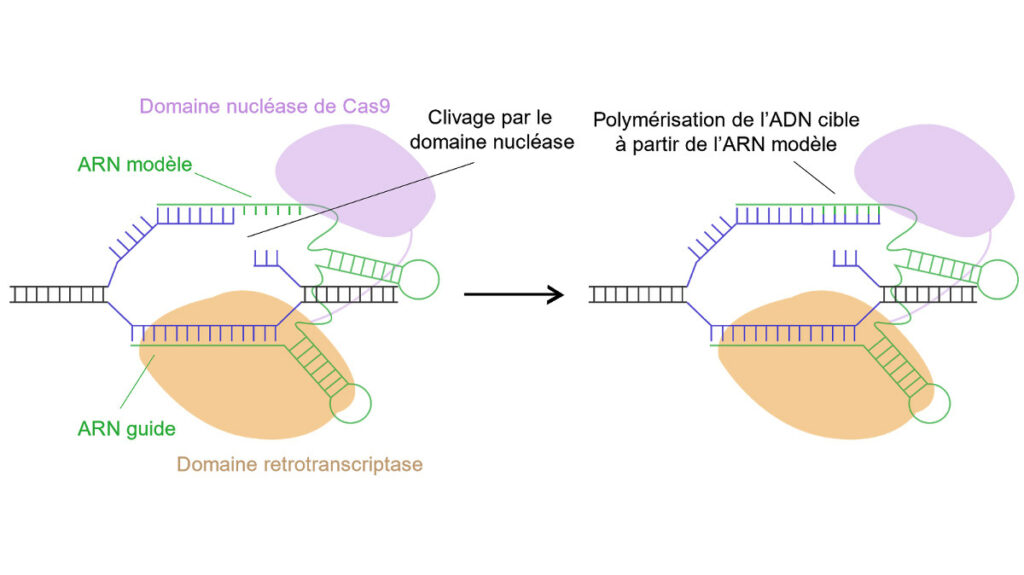
Cette méthode permet ainsi de chercher n’importe quelle séquence dans un génome et de la remplacer par une séquence voulue sans créer de cassures doubles brins, ni subir les restrictions de mutations possibles des éditeurs de bases. Cette méthode est encore très récente mais a déjà montré une bonne efficacité dans des bactéries et levures comme dans des lignées humaines.
Ces nouvelles technologies basées sur le système CRISPR-Cas9 permettent donc de nouvelles éditions du génome en supprimant les inconvénients les plus importants de la méthode originelle. Différentes approches du système ont permis de mettre au point des outils divers basés sur ce système, montrant le très large potentiel de CRISPR-Cas9. Il existe cependant encore des limites à ce système qui le rendent encore perfectible dans les années à venir.
Références
- Anzalone, A.V., Randolph, P.B., Davis, J.R., Sousa, A.A., Koblan, L.W., Levy, J.M., Chen, P.J., Wilson, C., Newby, G.A., Raguram, A., et al. (2019). Search-and-replace genome editing without double-strand breaks or donor DNA. Nature 576, 149–157.
- Brunner, E., Yagi, R., Debrunner, M., Beck-Schneider, D., Burger, A., Escher, E., Mosimann, C., Hausmann, G., and Basler, K. (2019). CRISPR-induced double-strand breaks trigger recombination between homologous chromosome arms. Life Sci. Alliance 2, e201800267.
- Jinek, M., Chylinski, K., Fonfara, I., Hauer, M., Doudna, J.A., and Charpentier, E. (2012). A Programmable Dual-RNA-Guided DNA Endonuclease in Adaptive Bacterial Immunity. Science 337, 816–821.
- Landrum, M.J., Lee, J.M., Riley, G.R., Jang, W., Rubinstein, W.S., Church, D.M., and Maglott, D.R. (2014). ClinVar: public archive of relationships among sequence variation and human phenotype. Nucleic Acids Res. 42, D980–D985.
- Qi, L.S., Larson, M.H., Gilbert, L.A., Doudna, J.A., Weissman, J.S., Arkin, A.P., and Lim, W.A. (2013). Repurposing CRISPR as an RNA-Guided Platform for Sequence-Specific Control of Gene Expression. Cell 152, 1173–1183.